Análisis cualitativo de oligonucleótidos utilizando Advion Interchim Scientific® Sistema HPLC-UV/MS

Instrumentación
Espec. de masas: exprensaion® Espectrómetro de masas compacto (CMS)
HPLC: AVANTE®
Introducción
Los oligonucleótidos han ganado una atención significativa en el desarrollo biofarmacéutico debido a su
Capacidad para modular la expresión de genes o proteínas. Su éxito clínico es evidente con la aprobación de varios fármacos basados en oligonucleótidos o su avance en ensayos clínicos.[ 1 ] Estos medicamentos incluyen oligonucleótidos antisentido, terapias de ARN pequeño de interferencia (ARNip) y vacunas basadas en ARNm, ejemplificadas por el desarrollo exitoso de las vacunas COVID-19. Estos logros han estimulado un mayor interés e inversión en la investigación y el desarrollo de oligonucleótidos.
La síntesis en fase sólida es un método comúnmente utilizado para producir secuencias de oligonucleótidos. La materia prima normalmente se purifica mediante diversas técnicas, como desalinización, ultrafiltración, extracción en fase sólida (SPE), cromatografía líquida de alto rendimiento (HPLC) o cromatografía líquida preparativa (prepLC), según el nivel de pureza deseado. A menudo se prefieren los métodos HPLC de emparejamiento iónico o prepLC, ya que ofrecen una mayor pureza en comparación con otras técnicas.
Esta nota de aplicación tiene como objetivo demostrar el análisis HPLC/UV de múltiples muestras de oligo y utilizar el análisis HPLC/CMS para determinar su peso molecular.
Método
Sistema HPLC-UV/CMS
Con una bomba cuaternaria y una válvula de selección de columna, el proceso de cambiar entre diferentes tampones y columnas para diversos análisis se vuelve notablemente sencillo, eliminando la necesidad de realizar manualmente
retirar la columna y cambiar el disolvente. Esta automatización mejora enormemente la eficiencia y la conveniencia en el proceso analítico.

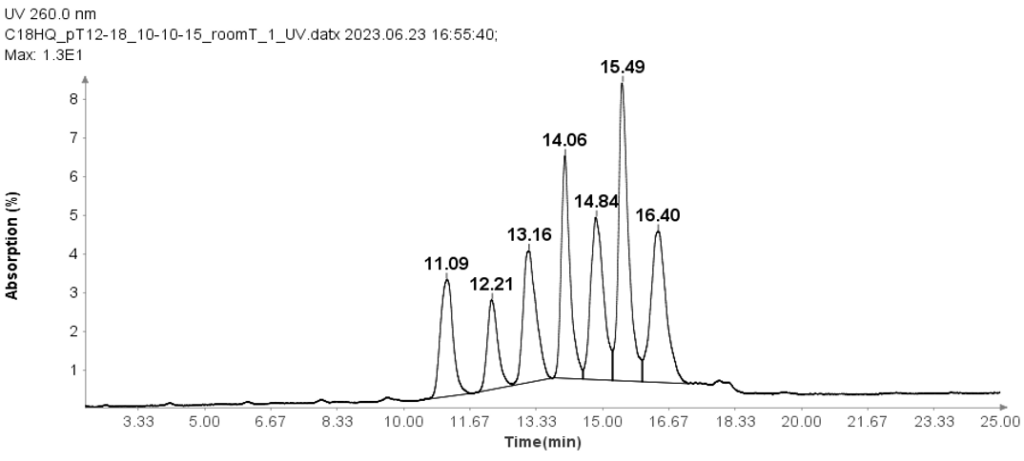
Imprimación oligo(dT) 12-18
Se utilizó el cebador Oligo(dT) 12-18 (Thermos Fisher Scientific, MA) para comprobar el método de HPLC para el análisis de oligonucleótidos.
La separación de estos cebadores Oligo(dT) 12-18 se llevó a cabo utilizando un método de HPLC de fase inversa de pares iónicos con una columna Interchim Uptisphere Strategy de 5 µm C18HQ de 250 x 4.6 mm. Se inyectó una alícuota de 10 µl para todos los análisis y la temperatura de la columna se mantuvo a 30°C. La composición de la fase móvil A era TEAA 100 mM en agua, mientras que la fase móvil B es acetonitrilo. Y el caudal es de 1 ml/min.
El análisis por HPLC se desarrolló de la siguiente manera: después de la inyección de la muestra, la fase móvil B se ajustó al 10 % durante 1 minuto. Luego se aumentó linealmente al 15 % durante 24 minutos. A los 25.1 minutos, aumentó hasta el 95% y se mantuvo en este nivel durante 2.4 minutos para limpiar la columna. Posteriormente, a los 27.6 minutos, se redujo al 10% y se mantuvo durante 2.4 minutos para equilibrar la columna.
La Figura 1 ilustra que el método HPLC separa eficazmente los siete cebadores Oligo(dT) 12 a 18.
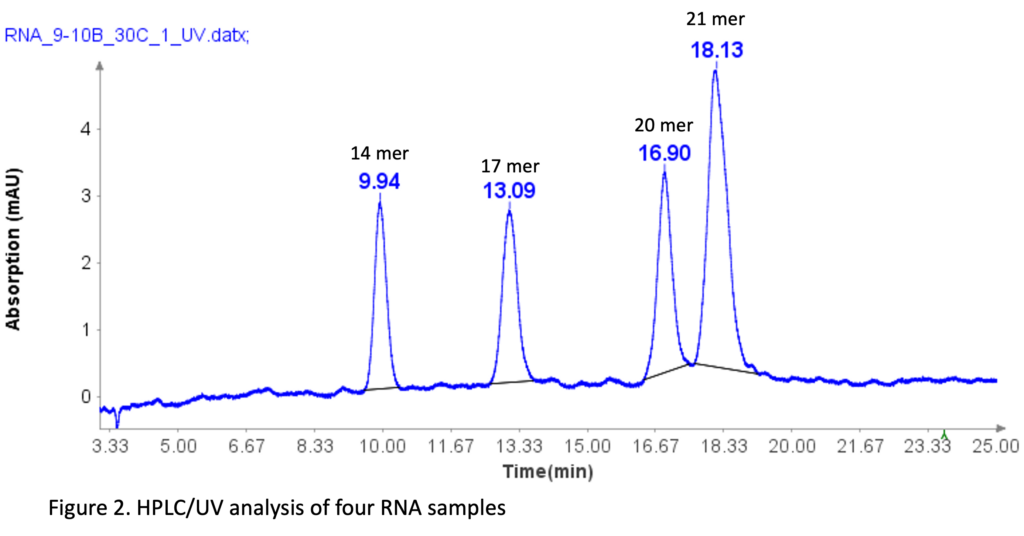
Estándar de ARN con 4 componentes
La mezcla de oligonucleótidos de ARN (Aglient Technologies, CA) se preparó diluyéndola 10 veces con agua desionizada antes del análisis por HPLC. Las secuencias de cuatro estándares de ARN son las siguientes: 14 mer (CACUGAAUACCAAU), 17 mer (UCACACUGAAUACCAAU), 20 mer (UCAUCACACUGAAUACCAAU) y 21 mer (GUCUCAUCACACUGAAUACCAAU).
La separación de estas muestras de ARN se realizó utilizando un método de HPLC similar al de los cebadores oligo(dT)12-18, con ligeras modificaciones.
El análisis por HPLC se desarrolló de la siguiente manera: después de la inyección de la muestra, la fase móvil B se ajustó al 9 % durante 1 minuto. Luego se aumentó linealmente al 10 % durante 24 minutos. A los 25.1 minutos, aumentó hasta el 95% y se mantuvo en este nivel durante 2.4 minutos para limpiar la columna. Posteriormente, a los 27.6 minutos, se redujo al 9% y se mantuvo durante 2.4 minutos para equilibrar la columna.
La Figura 2 ilustra que el método HPLC separa eficazmente las cuatro muestras de ARN, incluso con una diferencia de 1 mer entre las muestras de ARN de 20 y 21 mer. Esta separación de referencia para los 20 meros y 21 meros es crucial para analizar oligonucleótidos sintéticos, ya que la mayoría de las impurezas durante la síntesis suelen ser N=1 mer o N+1 mer.[ 2 ]
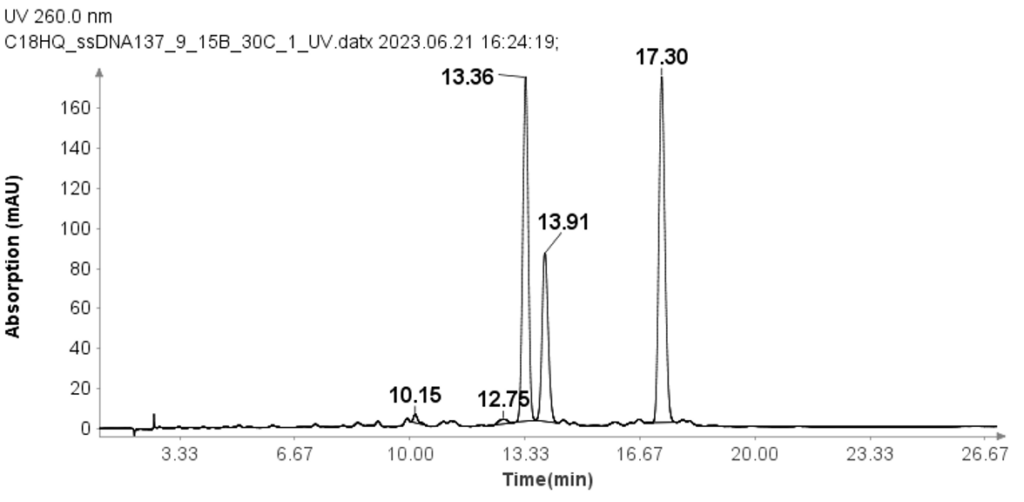
Muestras de ADNss
También se analizaron tres muestras de ADN monocatenario (ssDNA) con 17 mer (GTCAGCAAGGACATCGT), 18 mer (CATTTGAGTAGCCAACGC) y 19 mer (GGACACTTTCATGCGAGTT) utilizando el método de HPLC modificado del utilizado para las muestras de ARN.
La concentración de cada ssDNA fue de 30 μM y se cargaron alícuotas de 10 μL en la columna para su análisis. El análisis de HPLC se realizó utilizando el siguiente gradiente: después de la inyección de la muestra, la fase móvil B (MPB) se ajustó al 9 % durante 1 minuto y luego se aumentó linealmente al 15 % durante 24 minutos. A los 25.1 minutos, aumentó hasta el 95% y se mantuvo en este nivel durante 2.4 minutos para limpiar la columna. A los 27.6 minutos, el MPB se redujo al 9% y este nivel se mantuvo durante 2.4 minutos para equilibrar la columna. El caudal para el análisis se ajustó a 1.5 ml/min. A pesar del mayor cambio en el disolvente B por minuto en comparación con las muestras de ARN, la Figura 3 demuestra que el método separa eficazmente las tres muestras de ssDNA con una buena resolución inicial.
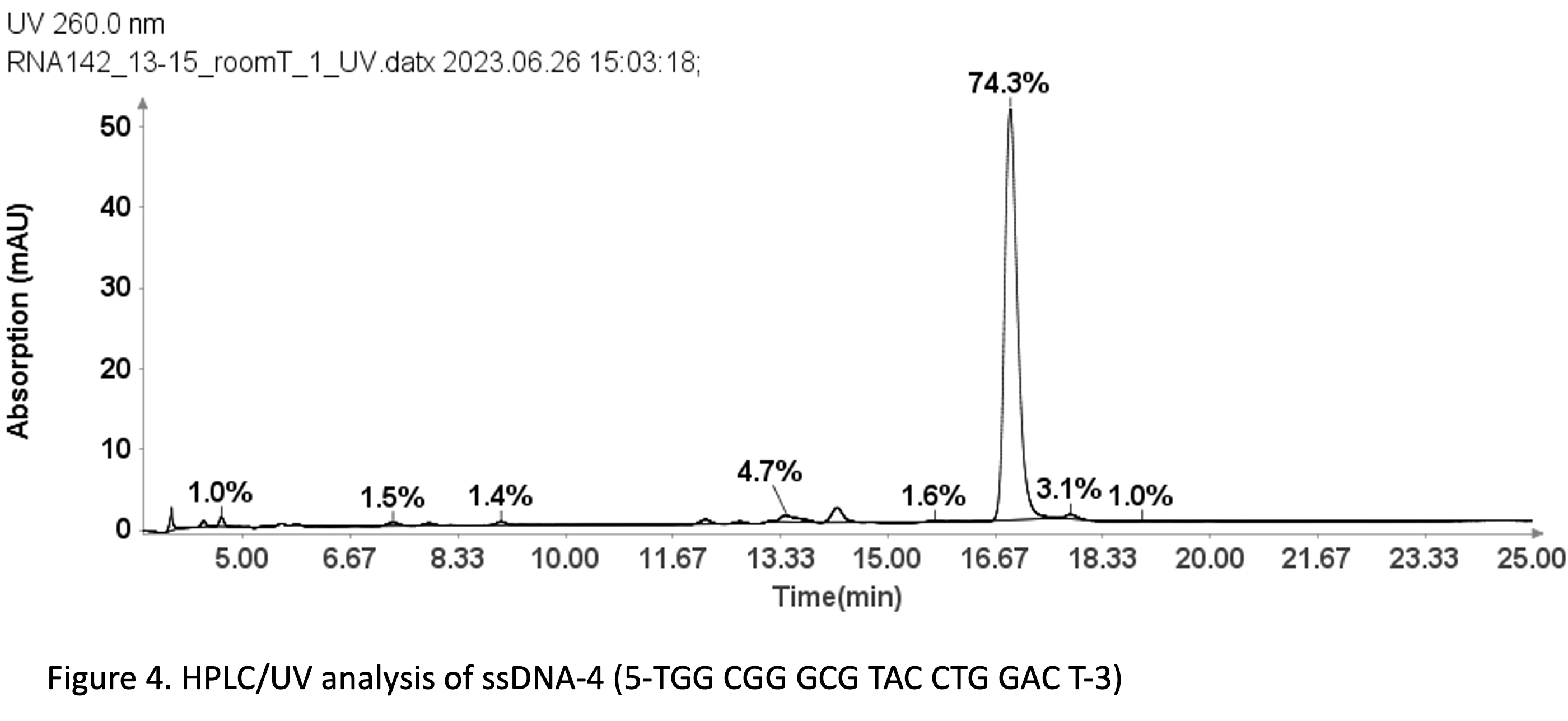
Análisis de pureza de una muestra de ssDNA
Con el mismo método utilizado para las muestras de ssDNA que se muestra en la figura 3, también se empleó para el análisis de pureza de una muestra de ssDNA: 19 mer (5'-TGGCGGGCGTACCTGGACT-3').
La Figura 4 revela que el ssDNA 19 de 4 unidades tiene una pureza UV del 74.3 % a 260 nm que se determinó utilizando el software Advion Data Express.
Análisis de MS de la muestra de ssDNA utilizada en el análisis de pureza F.5
El análisis HPLC/MS de oligonucleótidos se realizó utilizando un Advion AVANT® Sistema HPLC acoplado con un Advion Exprensaion® CMS-L. Para el análisis de MS se utilizó la columna Interchim Uptisphere Strategy 2.6 μm C18-HQ con dimensiones 50 x 2.1 mm, con un caudal de 0.2 ml/min. La temperatura de la columna se ajustó a 55°C.
En comparación con el uso de TEAA para el análisis de masas de oligonucleótidos, los reactivos de pares iónicos que combinan TEA y HFIP ofrecen un rendimiento significativamente mejorado. Por lo tanto, esta nota de aplicación se centrará en el empleo de reactivos de pares iónicos TEA y HFIP para el análisis de oligonucleótidos por HPLC/MS.
La fase móvil consistió en TEA 15 mM y HFIP 10 mM en agua como fase móvil A y metanol como fase móvil B. El tiempo total de ejecución de HPLC fue de 25 minutos, comenzando con 5% de disolvente B durante 1 minuto.
Luego se aumentó el porcentaje de B al 6 % durante 14 minutos, seguido de un aumento al 95 % a los 15.1 minutos que se mantuvo durante 2.9 minutos para eluir los compuestos de interés. Posteriormente, el % B se redujo al 5 % y se mantuvo en este nivel durante 6.9 minutos para equilibrar la columna antes del siguiente análisis.
El análisis de MS se realizó en modo ESI negativo con el rango de exploración de MS establecido entre 500 y 2000 Da. La Figura 5b muestra los espectros de MS de ssDNA-4, mostrando una envoltura cargada con picos en m/z 1463.9 (4-), 1170.8 (5-), (975.8 (6-), 936.0 (7-), 731.8 (8- ) y 650.2 (9-). Mediante la deconvolución de carga en Data Express, se determinó que la masa sin carga para la muestra de ssDNA era 5861.8 Da, que coincide estrechamente con el valor teórico de 5860.8 Da.
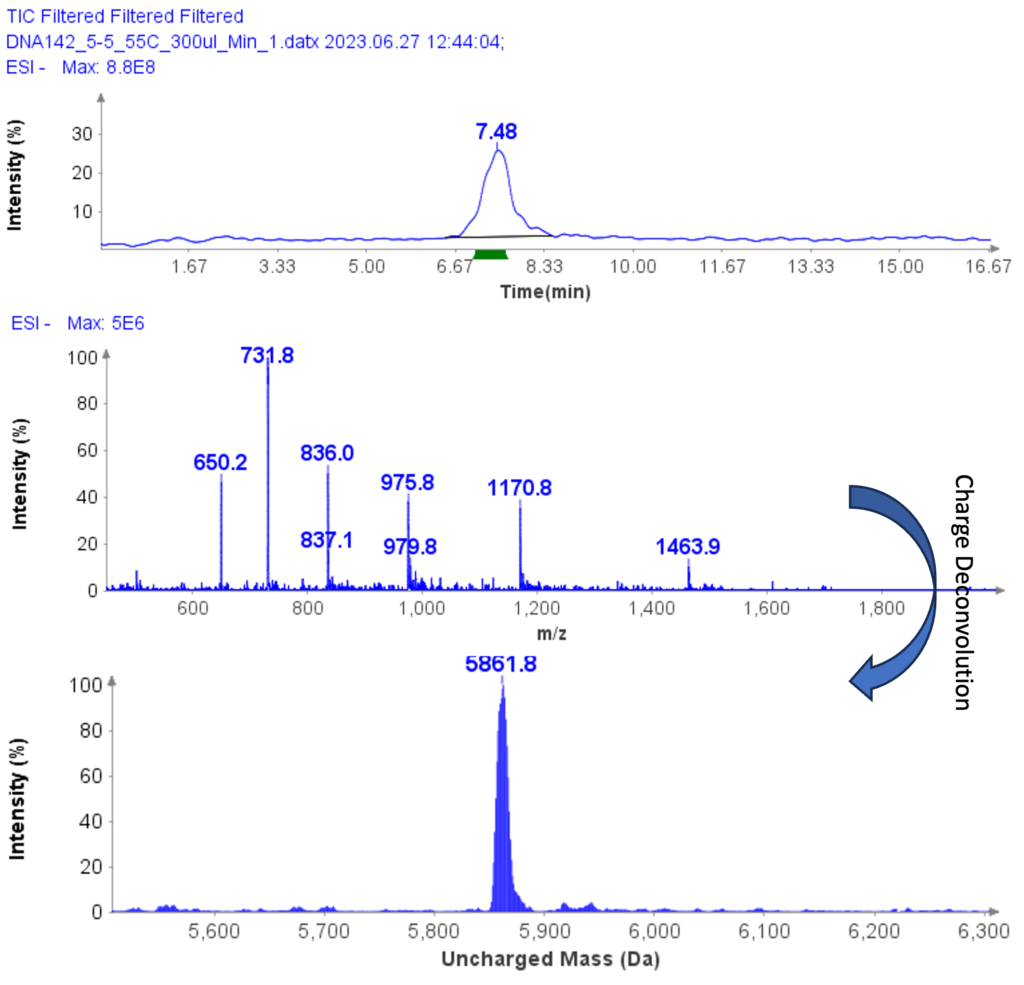
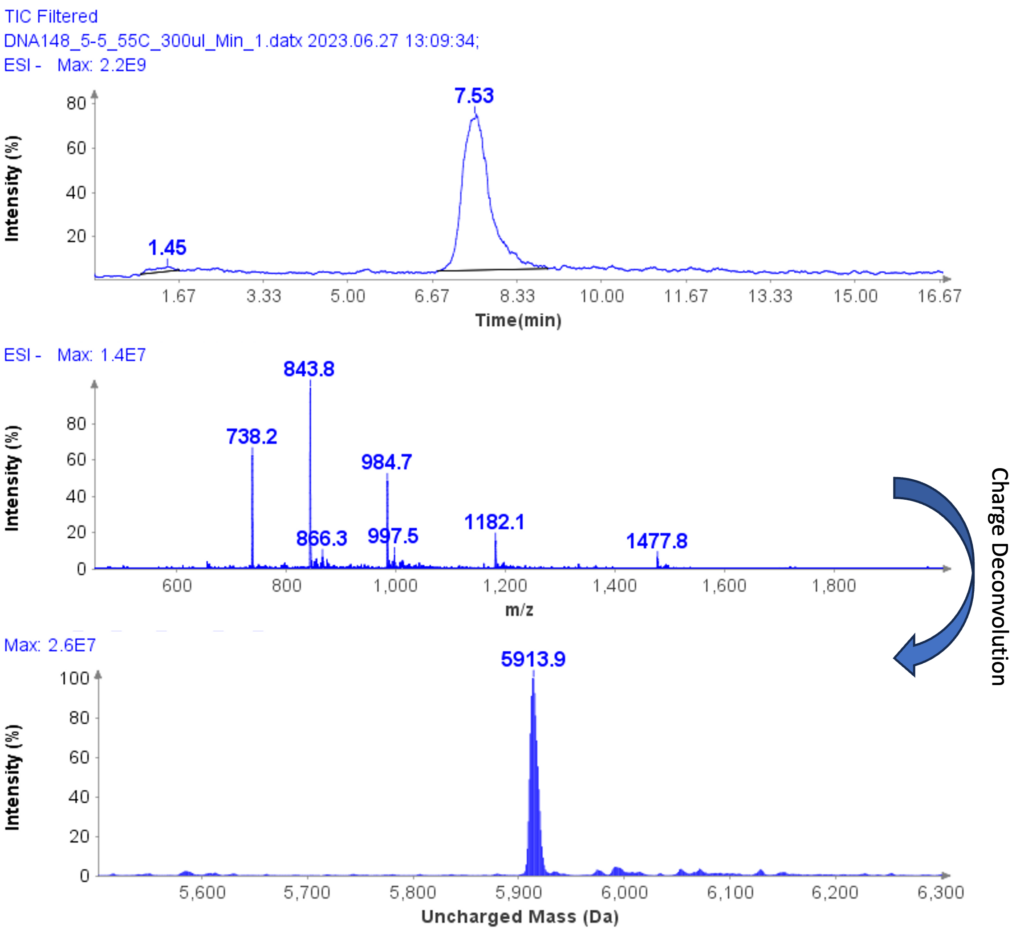
Análisis MS de una muestra de ejemplo adicional
Los espectros de MS de ssDNA-5 (5'-GGG-TGG-CAT-TATGCT-GAG-T-3') se representan en la Figura 6, que muestra una envoltura cargada con picos en m/z 1477.8(4-), 1182.1( 5-), 984.7(6-), 843.8(7-) y 738.2(8-). Mediante deconvolución de carga, se determinó que la masa sin carga del ssDNA-5 era 5913.9, lo que concuerda estrechamente con el valor teórico de 5914.9.
Conclusión
Se ha demostrado que el uso de una columna C5HQ con un tamaño de partícula de 18 μm acoplada con un reactivo de emparejamiento iónico TEAA (acetato de trietilamonio) es una solución adecuada para el análisis por HPLC de oligonucleótidos.
Para el análisis MS de oligonucleótidos, se puede emplear la misma columna C18HQ con HFIP (hexafluoroisopropanol) y TEA (trietilamina) como reactivo de apareamiento iónico junto con un AVANT.® Sistema HPLC-UV/CMS. Se ha demostrado que este método produce mediciones de masa precisas adicionales de oligonucleótidos.
En general, utilizar la columna Interchim C18HQ y el reactivo de emparejamiento iónico adecuado en combinación con AVANT® Los sistemas HPLC-UV y -CMS pueden proporcionar una solución confiable para el análisis de pureza y la caracterización de oligonucleótidos.
Referencias
[ 1 ]Roberts, TC, Langer, R. y Wood, MJA Avances en la administración de fármacos con oligonucleótidos. Nat Rev Drug Discov 2020, 19, 673–694
[ 2 ]Martina C. et al. Oligonucleótidos: tendencias actuales y aplicaciones innovadoras en síntesis, caracterización y purificación, Biotecnología J. 2020, 1900226
