Análisis de superficie de extracción de líquidos Espectrometría de masas de patógenos ESKAPE
Autores: Universidad de Birmingham, Reino Unido
Resumen
Los patógenos ESKAPE (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa y Enterobacter cloacae) representan especies bacterianas clínicamente importantes que son responsables de la mayoría de las infecciones farmacorresistentes adquiridas en el hospital; por tanto, la necesidad de una identificación rápida es de gran importancia. Trabajos anteriores han demostrado la idoneidad de la espectrometría de masas de análisis de superficie de extracción de líquidos (LESA-MS) para el análisis directo de colonias de dos de los patógenos ESKAPE (Staphylococcus aureus y Pseudomonas aeruginosa) creciendo en agar. Aquí, aplicamos LESA-MS a las cuatro especies restantes de ESKAPE (E. faecium E745, K. pneumoniae KP257, A. baumannii AYE, y E. cloacae S11) así como E. faecalis V583 (un pariente cercano de E. faecium) y un aislado clínico de A. baumannii AC02 utilizando un sistema de muestreo de disolvente optimizado. En cada caso, se empleó LESA MS / MS de arriba hacia abajo para la identificación de proteínas. En total, se identificaron 24 proteínas a partir de 37 espectros MS / MS mediante la búsqueda en bases de datos de proteínas para las especies individuales. Los espectros MS / MS para las proteínas identificadas se buscaron posteriormente en múltiples bases de datos de múltiples especies en un flujo de trabajo de análisis de datos automatizado con el fin de determinar la precisión de la identificación de las incógnitas. De las 24 proteínas, 19 se asignaron correctamente a nivel de proteína y especie, lo que corresponde a una tasa de éxito de identificación del 79%.
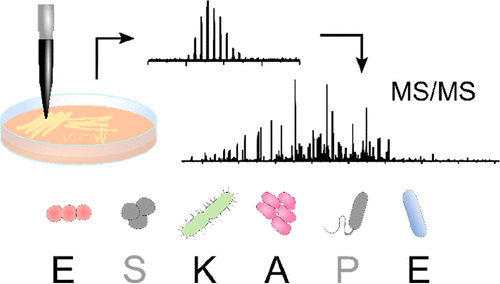
LESA-MS se realizó utilizando Advion Interchim Scientific® TriVersa NanoMate®.
