Analyse qualitative des oligonucléotides à l'aide du ® Système HPLC-UV/MS de Advion Interchim Scientific

Fournisseurs d'instruments
Spécification de masse: expression CMS® Spectromètre de masse compact (CMS)
HPLC:AVANT®
Introduction
Les oligonucléotides ont suscité une attention considérable dans le développement biopharmaceutique en raison de leur
capacité à moduler l’expression d’un gène ou d’une protéine. Leur succès clinique est évident avec l’approbation de plusieurs médicaments à base d’oligonucléotides ou leur progression vers les essais cliniques. Ces médicaments comprennent des oligonucléotides antisens, des produits thérapeutiques à petits ARN interférents (siARN) et des vaccins à base d'ARNm, illustrés par le développement réussi des vaccins contre la COVID-19. De telles réalisations ont stimulé davantage d’intérêt et d’investissements dans la recherche et le développement des oligonucléotides.
La synthèse en phase solide est une méthode couramment utilisée pour produire des séquences oligonucléotidiques. La matière première est généralement purifiée par diverses techniques, telles que le dessalage, l'ultrafiltration, l'extraction en phase solide (SPE), la chromatographie liquide haute performance (HPLC) ou la chromatographie liquide préparative (prepLC), en fonction du niveau de pureté souhaité. Les méthodes HPLC ou prepLC par appariement d'ions sont souvent préférées car elles offrent une pureté plus élevée que les autres techniques.
Cette note d'application vise à démontrer l'analyse HPLC/UV de plusieurs échantillons d'oligo et à utiliser l'analyse HPLC/CMS pour déterminer leur poids moléculaire.
Méthode
Système HPLC-UV/CMS
Avec une pompe quaternaire et une vanne de sélection de colonne, le processus de commutation entre différents tampons et colonnes pour diverses analyses devient remarquablement simple, éliminant le besoin de procéder manuellement.
retirer la colonne et changer le solvant. Cette automatisation améliore considérablement l’efficacité et la commodité du processus analytique.

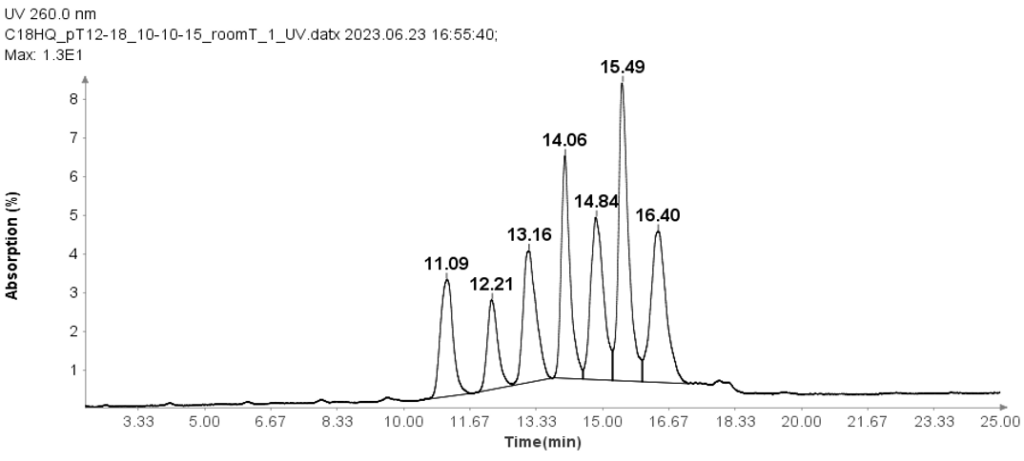
Amorce Oligo(dT) 12-18
L'amorce Oligo (dT) 12-18 (Thermos Fisher Scientific, MA) a été utilisée pour vérifier la méthode HPLC pour l'analyse des oligonucléotides.
La séparation de ces amorces Oligo (dT) 12-18 a été réalisée à l'aide d'une méthode HPLC en phase inverse par paire d'ions avec une colonne Interchim Uptisphere Strategy 5 µm C18HQ 250 x 4.6 mm. Une aliquote de 10 μL a été injectée pour toutes les analyses et la température de la colonne a été maintenue à 30 °C. La composition de la phase mobile A était de 100 mM de TEAA dans l'eau, tandis que la phase mobile B est de l'acétonitrile. Et le débit est de 1 ml/min.
L'analyse HPLC s'est déroulée comme suit : Après injection de l'échantillon, la phase mobile B a été fixée à 10 % pendant 1 minute. Elle a ensuite été augmentée linéairement jusqu'à 15 % sur 24 minutes. A 25.1 minutes, il est passé à 95 % et maintenu à ce niveau pendant 2.4 minutes pour nettoyer la colonne. Ensuite, à 27.6 minutes, elle a été réduite à 10 % et maintenue pendant 2.4 minutes pour l'équilibrage de la colonne.
La figure 1 montre que la méthode HPLC sépare efficacement les sept amorces Oligo(dT) 12 à 18.
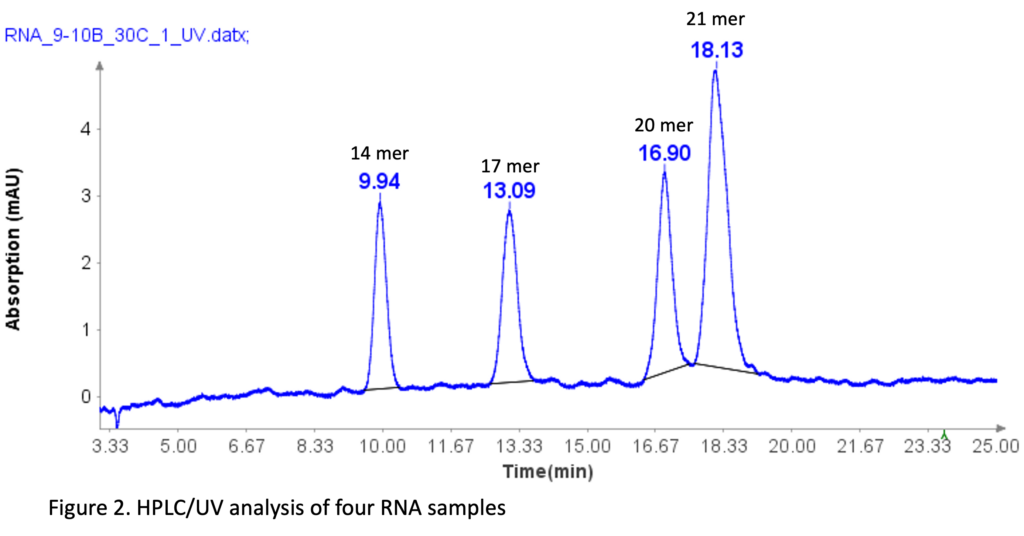
Standard ARN à 4 composants
Le mélange d'oligonucléotides d'ARN (Aglient Technologies, CA) a été préparé en le diluant 10 fois avec de l'eau DI avant analyse HPLC. Les séquences de quatre normes d'ARN sont les suivantes : 14 mers (CACUGAAUACCAAU), 17 mers (UCACACUGAAUACCAAU), 20 mers (UCAUCACACUGAAUACCAAU) et 21 mers (GUCUCAUCACACUGAAUACCAAU).
La séparation de ces échantillons d'ARN a été réalisée en utilisant une méthode HPLC similaire à celle des amorces oligo (dT) 12-18, avec de légères modifications.
L'analyse HPLC s'est déroulée comme suit : Après injection de l'échantillon, la phase mobile B a été fixée à 9 % pendant 1 minute. Elle a ensuite été augmentée linéairement jusqu'à 10 % sur 24 minutes. A 25.1 minutes, il est passé à 95 % et maintenu à ce niveau pendant 2.4 minutes pour nettoyer la colonne. Ensuite, à 27.6 minutes, elle a été réduite à 9 % et maintenue pendant 2.4 minutes pour l'équilibrage de la colonne.
La figure 2 montre que la méthode HPLC sépare efficacement les quatre échantillons d'ARN, même avec une différence de 1-mer entre les échantillons d'ARN 20-mer et 21-mer. Cette séparation de base pour les 20-mer et 21-mer est cruciale pour l'analyse des oligonucléotides synthétiques, car la plupart des impuretés lors de la synthèse sont généralement N=1 mer ou N+1 mer.
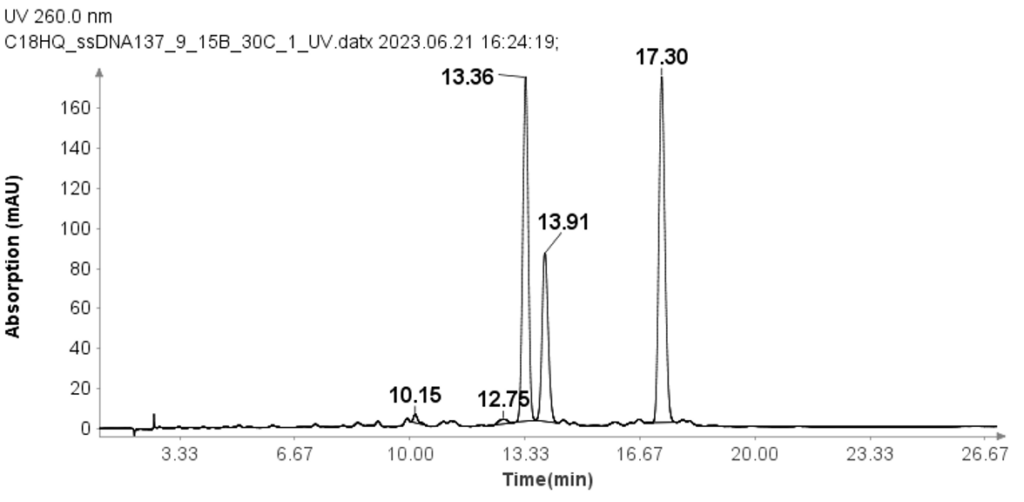
Échantillons d'ADNsb
Trois échantillons d'ADN simple brin (ADNsb) avec 17 mers (GTCAGCAAGGACATCGT), 18 mers (CATTTGAGTAGCCAACGC) et 19 mers (GGACACTTTCATGCGAGTT) ont également été testés en utilisant la méthode HPLC modifiée par rapport à celle utilisée pour les échantillons d'ARN.
La concentration de chaque ADNsb était de 30 µM et des aliquotes de 10 µL ont été chargées sur la colonne pour analyse. L'analyse HPLC a été réalisée en utilisant le gradient suivant : Après l'injection de l'échantillon, la phase mobile B (MPB) a été fixée à 9 % pendant 1 minute, puis augmentée linéairement jusqu'à 15 % sur 24 minutes. A 25.1 minutes, il est passé à 95 % et maintenu à ce niveau pendant 2.4 minutes pour nettoyer la colonne. Au bout de 27.6 minutes, le MPB a été réduit à 9 %, et ce niveau a été maintenu pendant 2.4 minutes pour l'équilibrage de la colonne. Le débit pour l'analyse a été réglé à 1.5 ml/min. Malgré le changement plus important du solvant B par minute par rapport aux échantillons d’ARN, la figure 3 démontre que la méthode sépare efficacement les trois échantillons d’ADN double brin avec une bonne résolution de base.
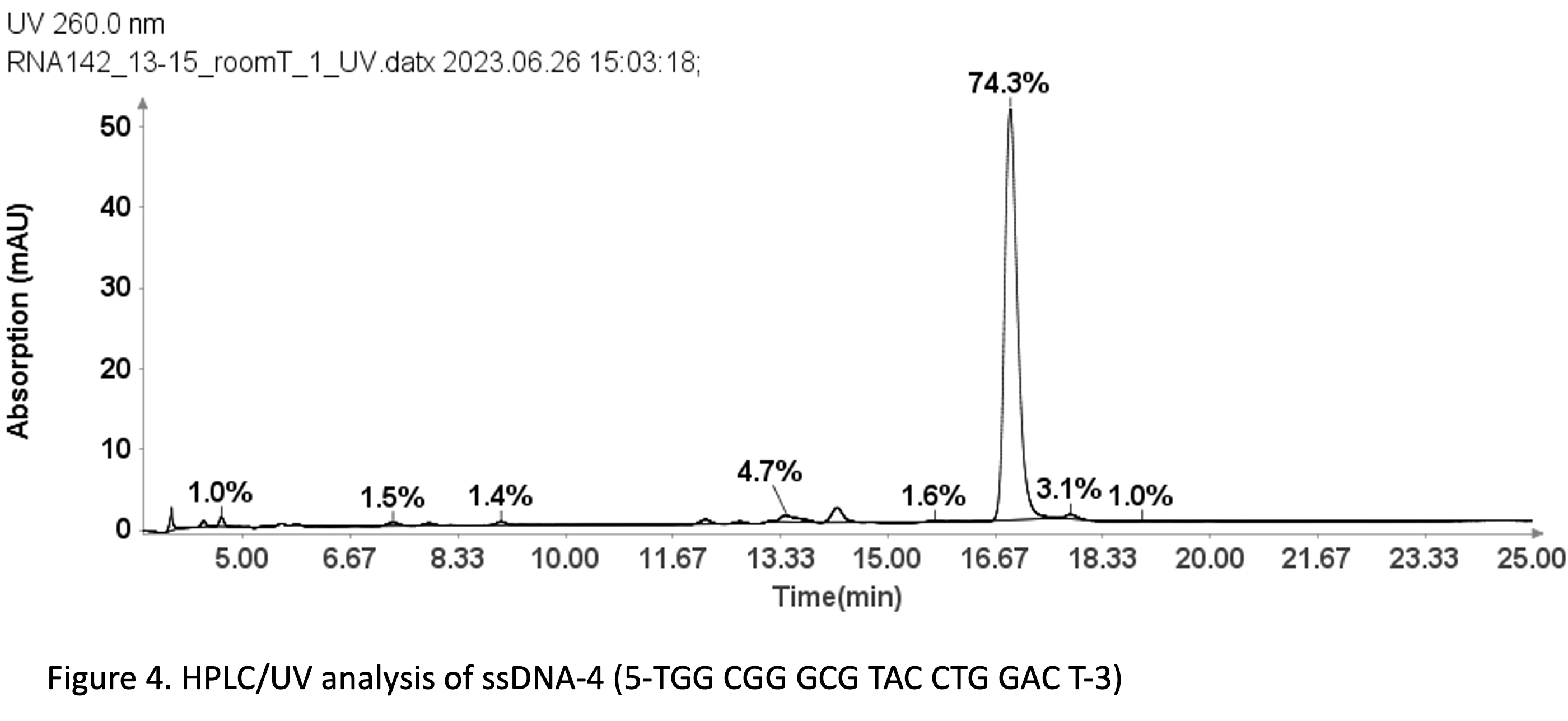
Analyse de pureté d'un échantillon d'ADNsb
Avec une même méthode utilisée pour les échantillons d'ADNsb présentée dans la figure 3, elle a également été utilisée pour l'analyse de pureté d'un échantillon d'ADNsb : 19 mer (5'-TGGCGGGCGTACCTGGACT-3').
La figure 4 révèle que l'ADNsb 19 de 4 mers a une pureté UV de 74.3 % à 260 nm, déterminée à l'aide du logiciel Advion Data Express.
Analyse MS de l'échantillon d'ADNsb utilisé dans l'analyse de pureté F.5
L'analyse HPLC/MS des oligonucléotides a été réalisée à l'aide d'un Advion AVANT® Système HPLC couplé à un Advion Expression CMS® CMS-L. Pour l'analyse MS, la colonne Interchim Uptisphere Strategy 2.6 µm C18-HQ de dimensions 50 x 2.1 mm a été utilisée, avec un débit de 0.2 ml/min. La température de la colonne a été réglée à 55°C.
Par rapport à l’utilisation du TEAA pour l’analyse de masse des oligonucléotides, les réactifs à paires d’ions combinant TEA et HFIP offrent des performances considérablement améliorées. Par conséquent, cette note d’application se concentrera sur l’utilisation des réactifs de paires d’ions TEA et HFIP pour l’analyse HPLC/MS des oligonucléotides.
La phase mobile était constituée de 15 mM de TEA et de 10 mM de HFIP dans de l'eau en tant que phase mobile A et de méthanol en tant que phase mobile B. La durée totale d'exécution de la HPLC était de 25 minutes, en commençant avec 5 % de solvant B pendant 1 minute.
Le pourcentage de B a ensuite été augmenté à 6 % sur 14 minutes, suivi d'une augmentation à 95 % à 15.1 minutes qui a été maintenue pendant 2.9 minutes pour éluer les composés d'intérêt. Par la suite, le % B a été réduit à 5 % et maintenu à ce niveau pendant 6.9 minutes pour équilibrer la colonne avant l'analyse suivante.
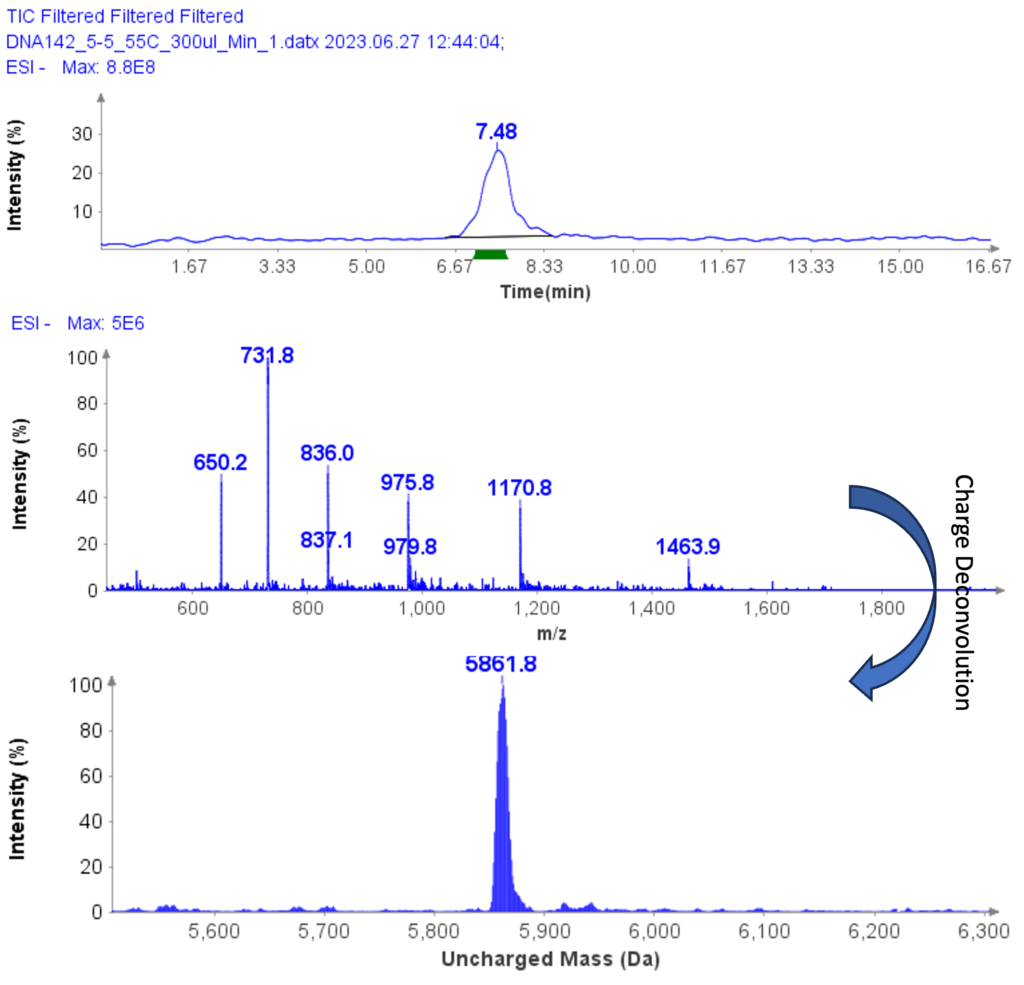
L'analyse MS a été réalisée en mode ESI négatif avec la plage de numérisation MS définie entre 500 et 2000 5 Da. La figure 4b montre les spectres MS de l'ADNsb-1463.9, affichant une enveloppe chargée avec des pics à m/z 4 (1170.8-), 5 (975.8-), (6 (936.0-), 7 (731.8-), 8 (650.2-). ) et 9 (5861.8-). Grâce à la déconvolution de charge dans Data Express, la masse non chargée de l'échantillon d'ADN simple brin a été déterminée comme étant de 5860.8 XNUMX Da, ce qui correspond étroitement à la valeur théorique de XNUMX XNUMX Da.
Analyse MS d'un autre exemple d'échantillon
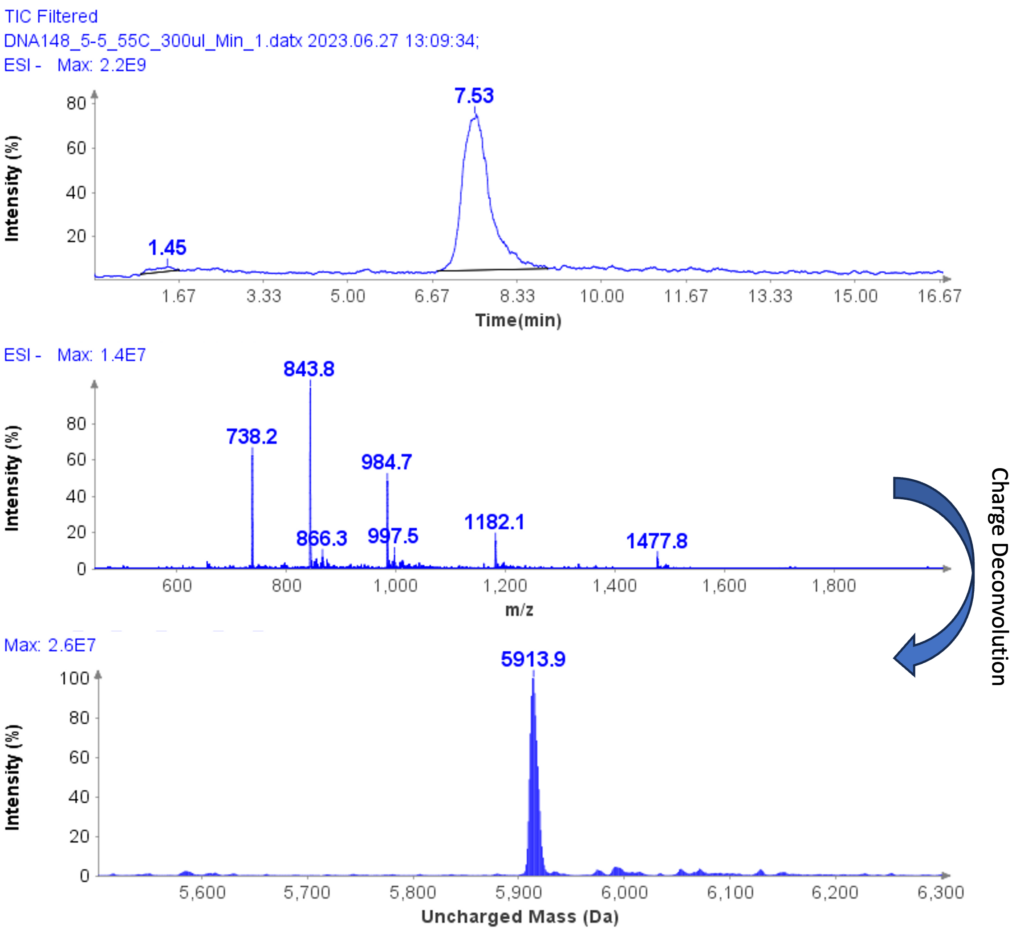
Les spectres MS de l'ADNsb-5 (5'-GGG-TGG-CAT-TATGCT-GAG-T-3') sont représentés sur la figure 6, montrant une enveloppe chargée avec des pics à m/z 1477.8(4-), 1182.1( 5-), 984.7(6-), 843.8(7-) et 738.2(8-). Par déconvolution de charge, la masse non chargée de l'ADNsb-5 a été déterminée à 5913.9, ce qui est en accord étroit avec la valeur théorique de 5914.9.
Conclusion
L’utilisation d’une colonne C5HQ de taille de particules de 18 µm couplée au réactif d’appariement d’ions TEAA (acétate de triéthylammonium) s’est avérée être une solution appropriée pour l’analyse HPLC des oligonucléotides.
Pour l'analyse MS des oligonucléotides, la même colonne C18HQ peut être utilisée avec HFIP (hexafluoroisopropanol) et TEA (triéthylamine) comme réactif d'appariement d'ions en conjonction avec un AVANT® Système HPLC-UV/CMS. Il a été prouvé que cette méthode donne des mesures de masse supplémentaires et précises des oligonucléotides.
Dans l'ensemble, en utilisant la colonne Interchim C18HQ et le réactif d'appariement d'ions approprié en combinaison avec l'AVANT® Les systèmes HPLC-UV et -CMS peuvent fournir une solution fiable pour l'analyse de la pureté et la caractérisation des oligonucléotides.
Références
Roberts, TC, Langer, R. & Wood, MJA Progrès dans l'administration de médicaments oligonucléotidiques. Nat Rev Drug Discov2020, 19, 673-694
Martina C. et.al. Oligonucléotides : tendances actuelles et applications innovantes en matière de synthèse, de caractérisation et de purification, Biotechnology J. 2020, 1900226
